-
 国外
国外
- AI生命科学OpenBioMLOpenBioML旨在成为机器学习和生物学交叉领域的开放协作研究实验室。
爱站权重:
OpenBioML是一个面向生物医学领域的开源科研社区,致力于把机器学习技术与生物学研究深度结合。项目以去中心化、完全开放的方式组织,提供一系列代码仓库、模型实现和评估工具,供科研人员自由使用、复现和扩展。社区受EleutherAI、DeepSpeed等前沿开源项目启发,聚焦蛋白质语言模型、化学语言模型、基础脑模型等方向,并通过项目提案模板推动社区协作。
OpenBioML平台特点:
1、多仓库生态:
目前拥有10余个公开仓库,涵盖protein‑lm‑scaling、bio‑chem‑lm、foundation‑brain‑models、lm‑evaluation‑harness、gpt‑neox等,形成从模型训练、评估到应用的完整链路。
2、完全开放:
代码、数据、模型均采用宽松的开源许可证,任何人均可克隆、修改并提交PR;社区不设门槛,鼓励跨机构、跨学科合作。
3、统一提案与日志:
提供项目提案模板和实验日志(lab‑log),规范科研过程,提升可复现性与透明度。
4、与主流框架兼容:
基于PyTorch、DeepSpeed、HuggingFaceTransformers等生态,实现大规模分布式训练和高效推理。
5、评估基准:
维护lm‑evaluation‑harness分支,支持少样本、零样本以及专业生物医学基准(如蛋白质功能预测、化学性质回归)。
6、社区驱动的资源共享:
模型权重、数据集、实验脚本均可在组织的GitHub页面统一获取,降低重复工作成本。
OpenBioML典型应用场景:
1、蛋白质语言模型:
通过protein‑lm‑scaling训练大规模蛋白质序列模型,用于结构预测、功能注释等。
2、化学语言模型:
bio‑chem‑lm处理小分子SMILES序列,实现属性预测、反应路径生成。
3、基础脑模型:
foundation‑brain‑models训练神经影像或脑电信号的自监督模型。
4、模型评估与基准:
lm‑evaluation‑harness为生物医学任务提供统一评测框架。
5、大模型训练加速:
基于DeepSpeed的gpt‑neox实现多卡并行、显存优化。
6、跨模态检索:
将文本、序列、结构信息统一映射,实现文献‑分子、文献‑蛋白质检索。
7、智能问答:
基于BioMedGPT的生物医学问答系统,支持疾病‑基因‑药物查询。

数据统计
特别声明&浏览提醒
本站AI工具导航站提供的「OpenBioML」的相关内容都来源于网络,不保证外部链接的准确性和完整性。在2023年08月11日 08时27分35秒收录时,该网站上的内容都属于合规合法,后期网站的内容如出现违规,可以直接联系网站管理员(ai@ipkd.cn)进行删除,AI工具导航站不承担任何责任。在浏览网页时,请注意您的账号和财产安全,切勿轻信网上广告!


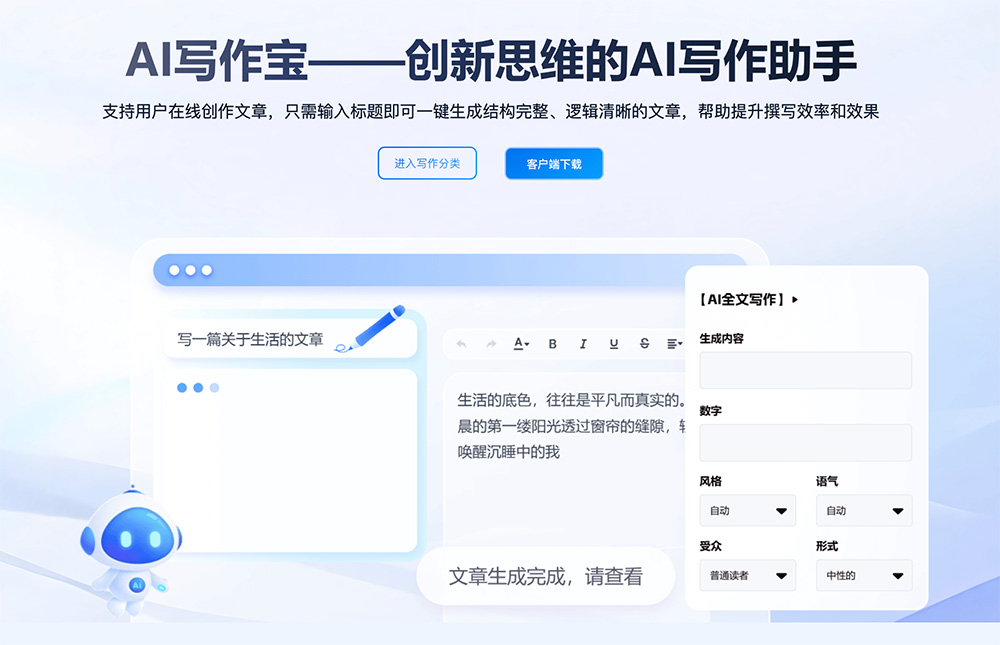 6款免费AI写作神器,一键生成文章、报告、公文、论文,高效又省心!
6款免费AI写作神器,一键生成文章、报告、公文、论文,高效又省心!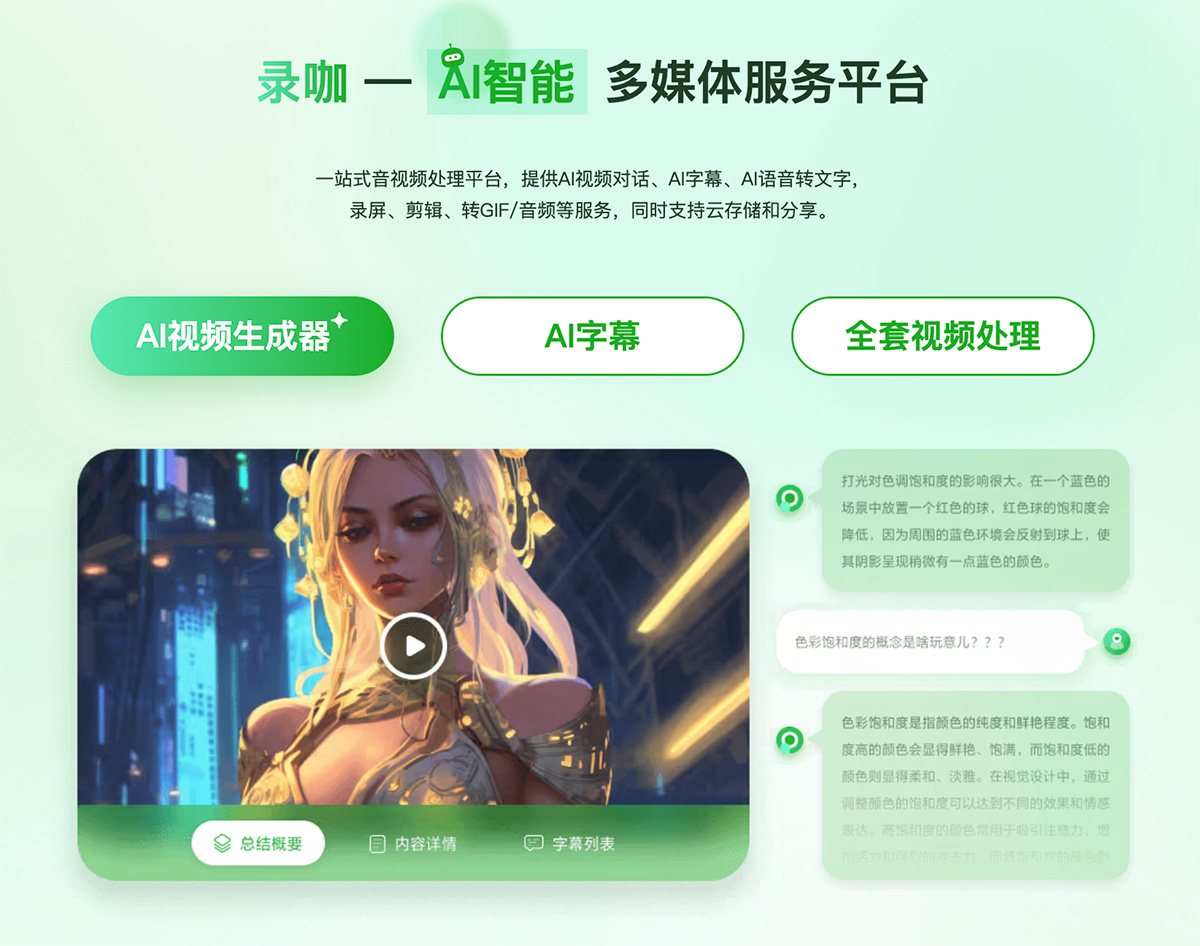 6款AI视频翻译与配音工具,一键生成多语言视频
6款AI视频翻译与配音工具,一键生成多语言视频 7款免费AI英语口语软件,一对一模拟对话,轻松开口说英语!
7款免费AI英语口语软件,一对一模拟对话,轻松开口说英语! 5个免费AI漫画生成工具,一键开启漫画创作之旅!
5个免费AI漫画生成工具,一键开启漫画创作之旅!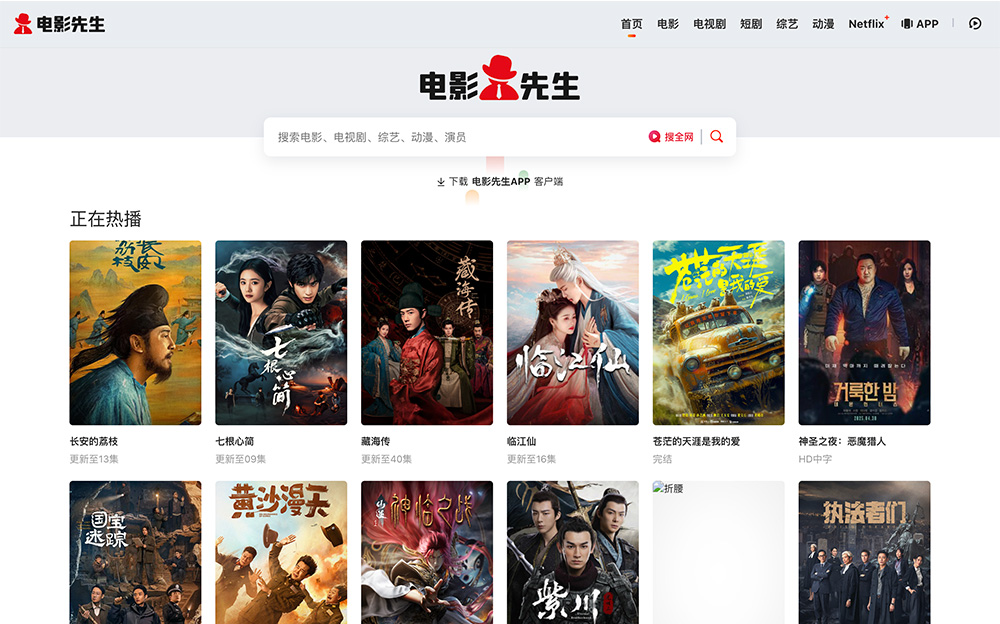 盘点8个欧美电影免费观看电视剧大全
盘点8个欧美电影免费观看电视剧大全 全能ai翻译软件哪个好用,这8个工具突破语言障碍
全能ai翻译软件哪个好用,这8个工具突破语言障碍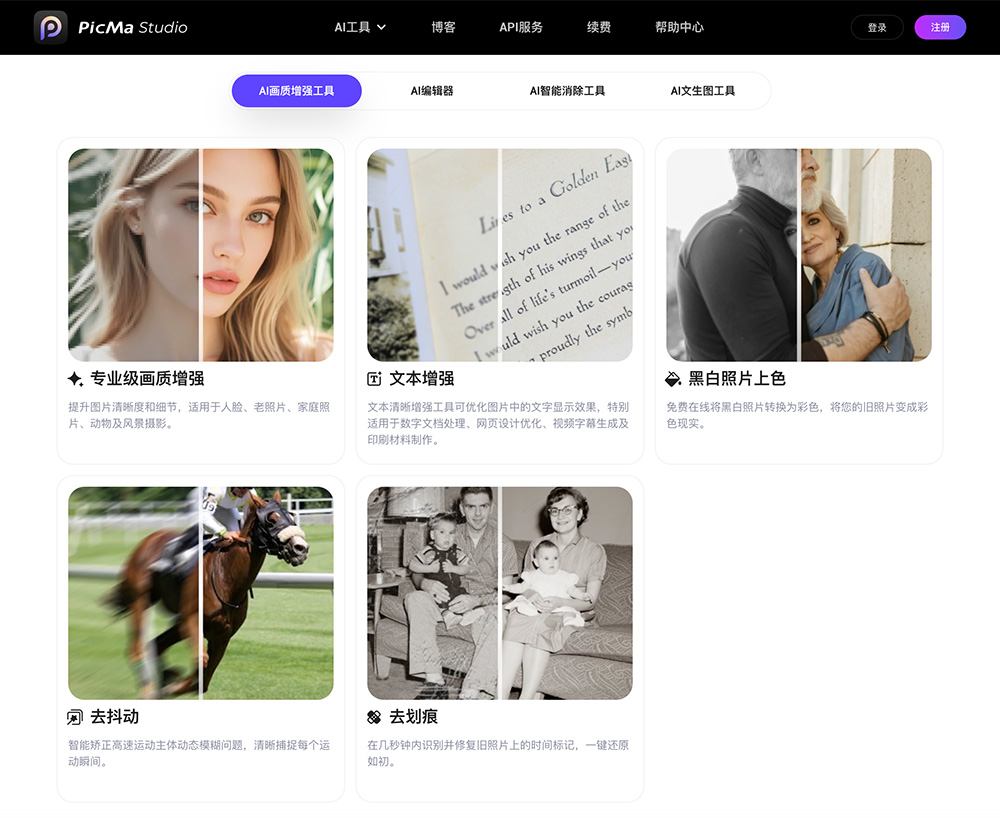 热门的ai老照片修复软件有哪些 2025高人气Ai绘图软件排行
热门的ai老照片修复软件有哪些 2025高人气Ai绘图软件排行 盘点7款主流AI训练模型:技术特性与应用场景解析
盘点7款主流AI训练模型:技术特性与应用场景解析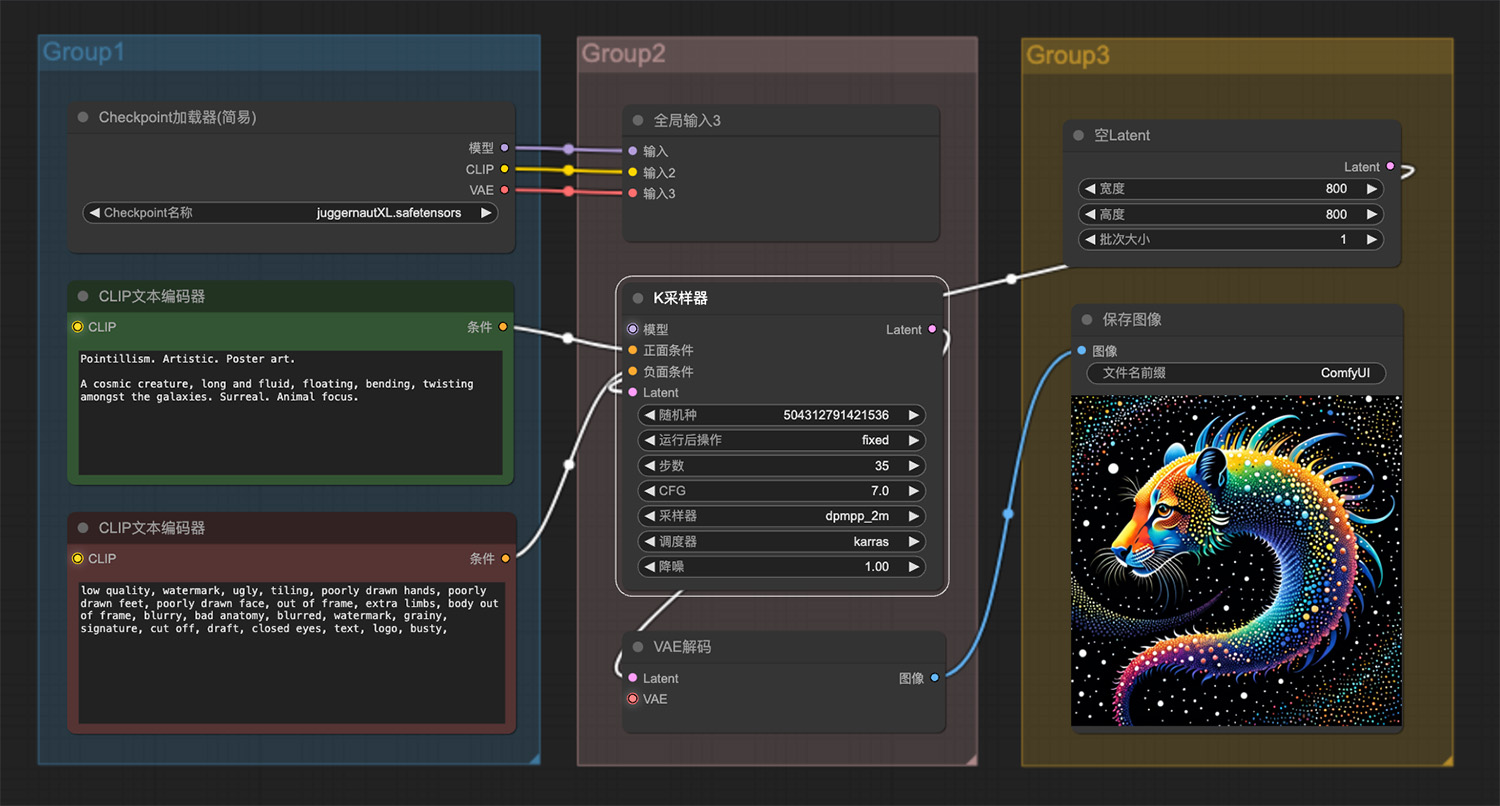 一只在星系中漂浮宇宙生物ComfyUI工作流
一只在星系中漂浮宇宙生物ComfyUI工作流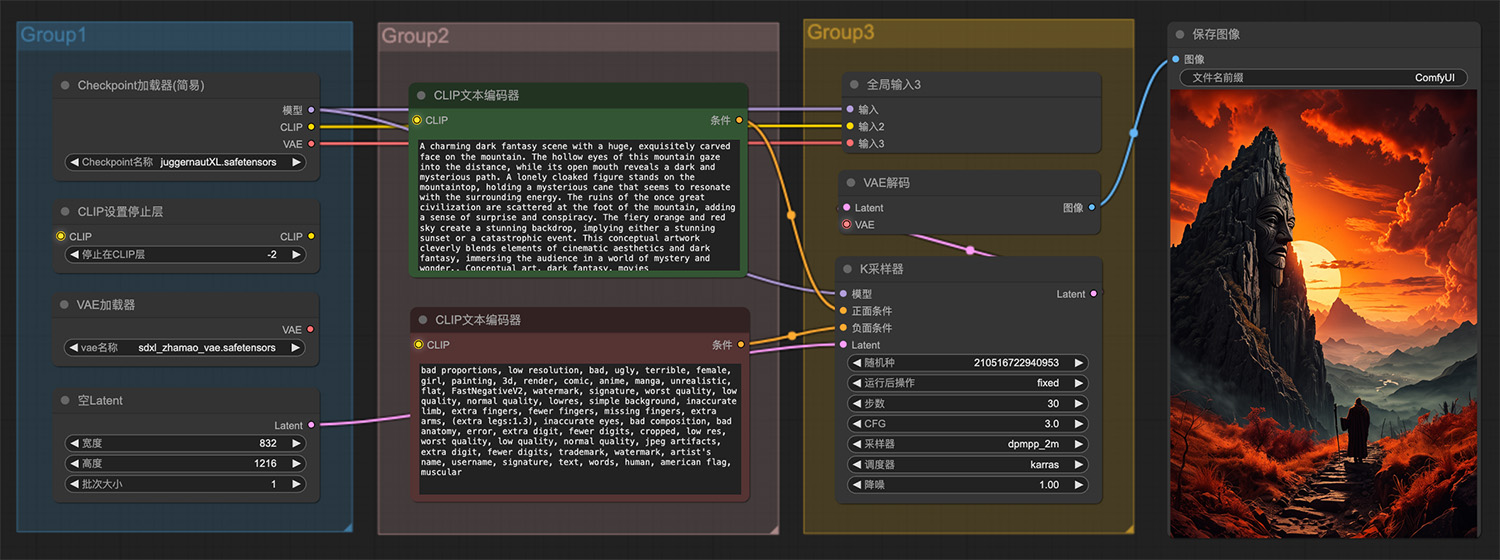 一个孤独的斗篷人物站在一座巨大的雕塑旁
一个孤独的斗篷人物站在一座巨大的雕塑旁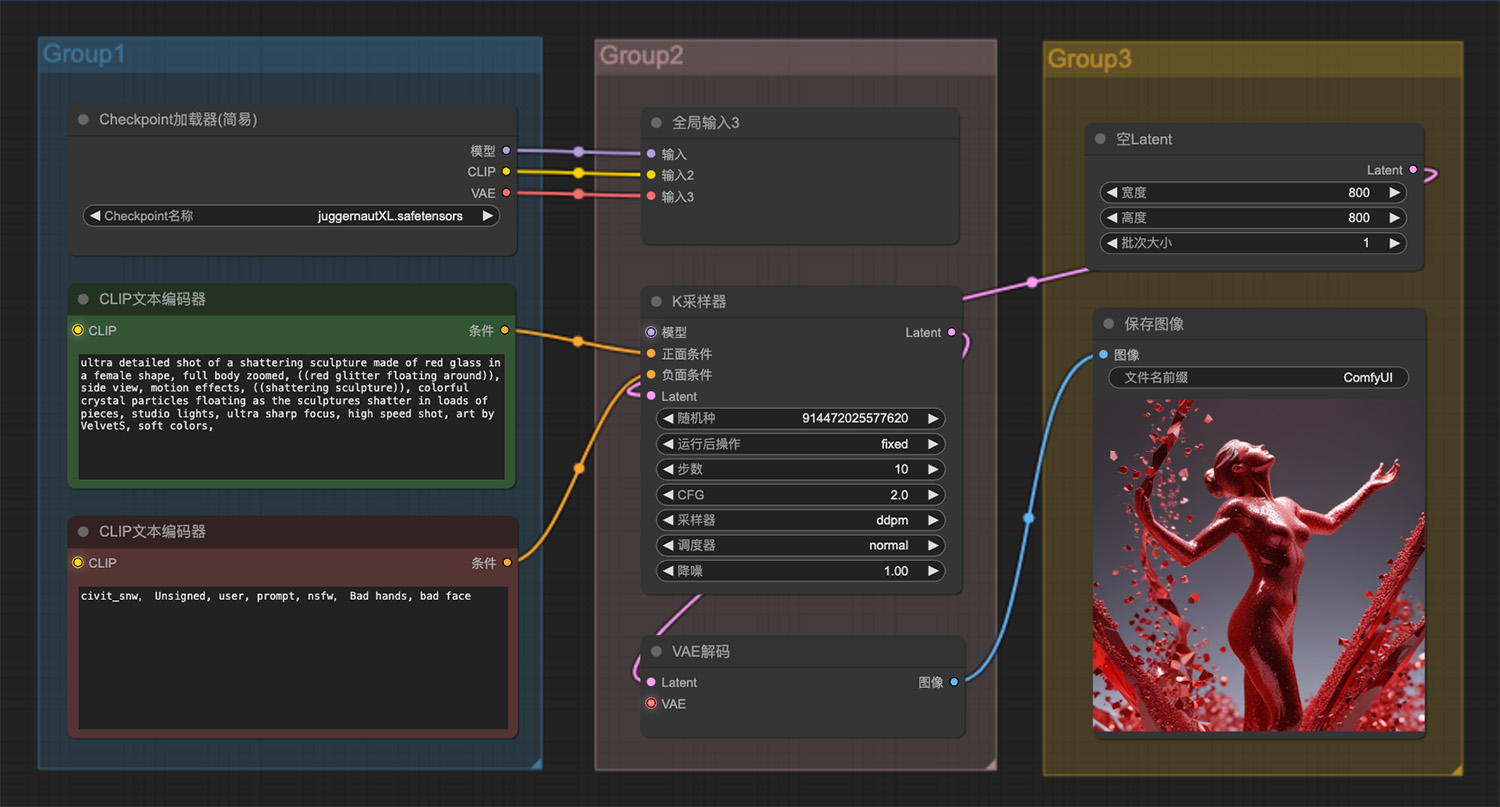 一个由红色玻璃制成的女性形状的破碎雕塑
一个由红色玻璃制成的女性形状的破碎雕塑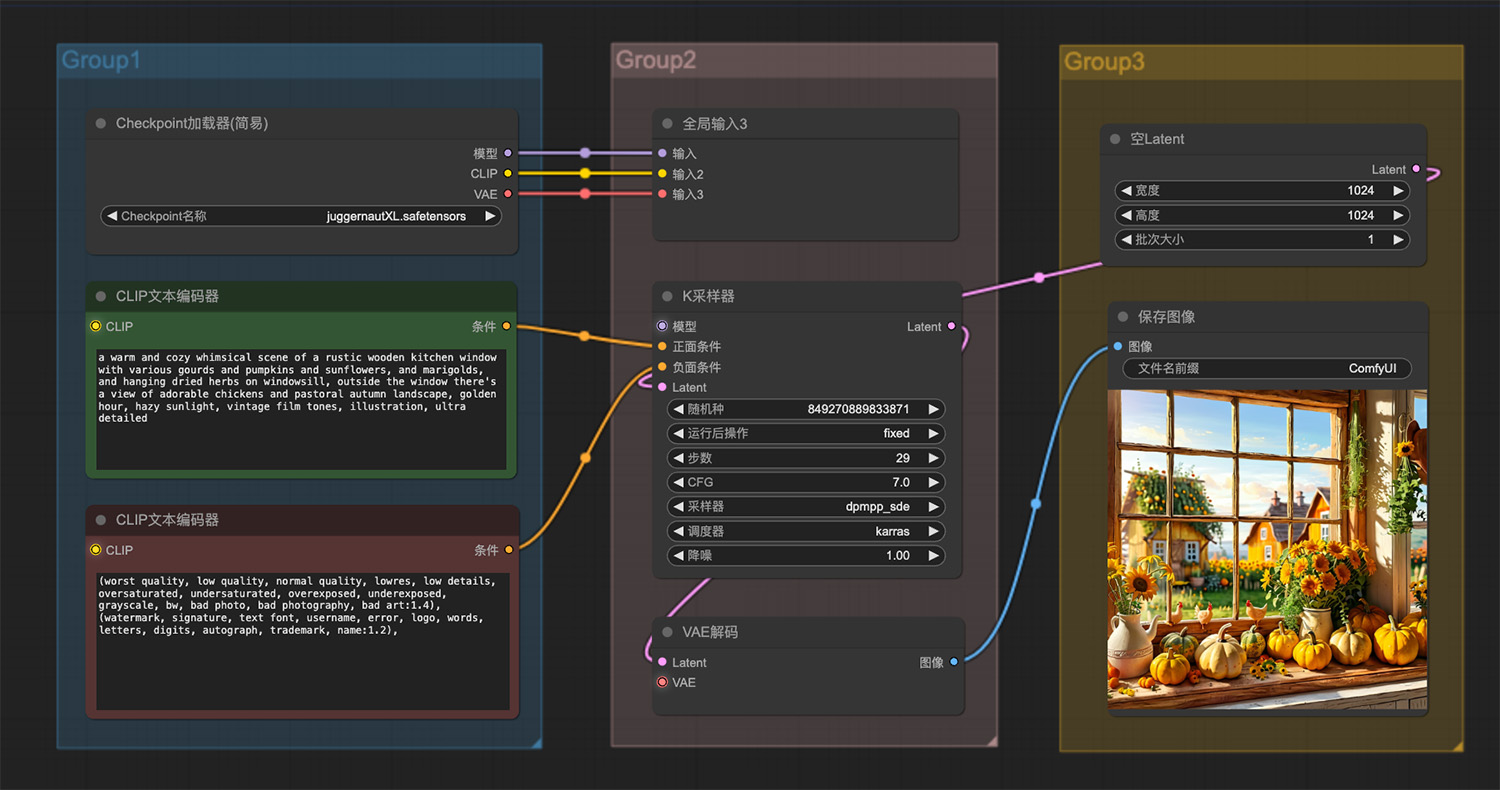 一个质朴的木制窗户上放着各种葫芦、南瓜等
一个质朴的木制窗户上放着各种葫芦、南瓜等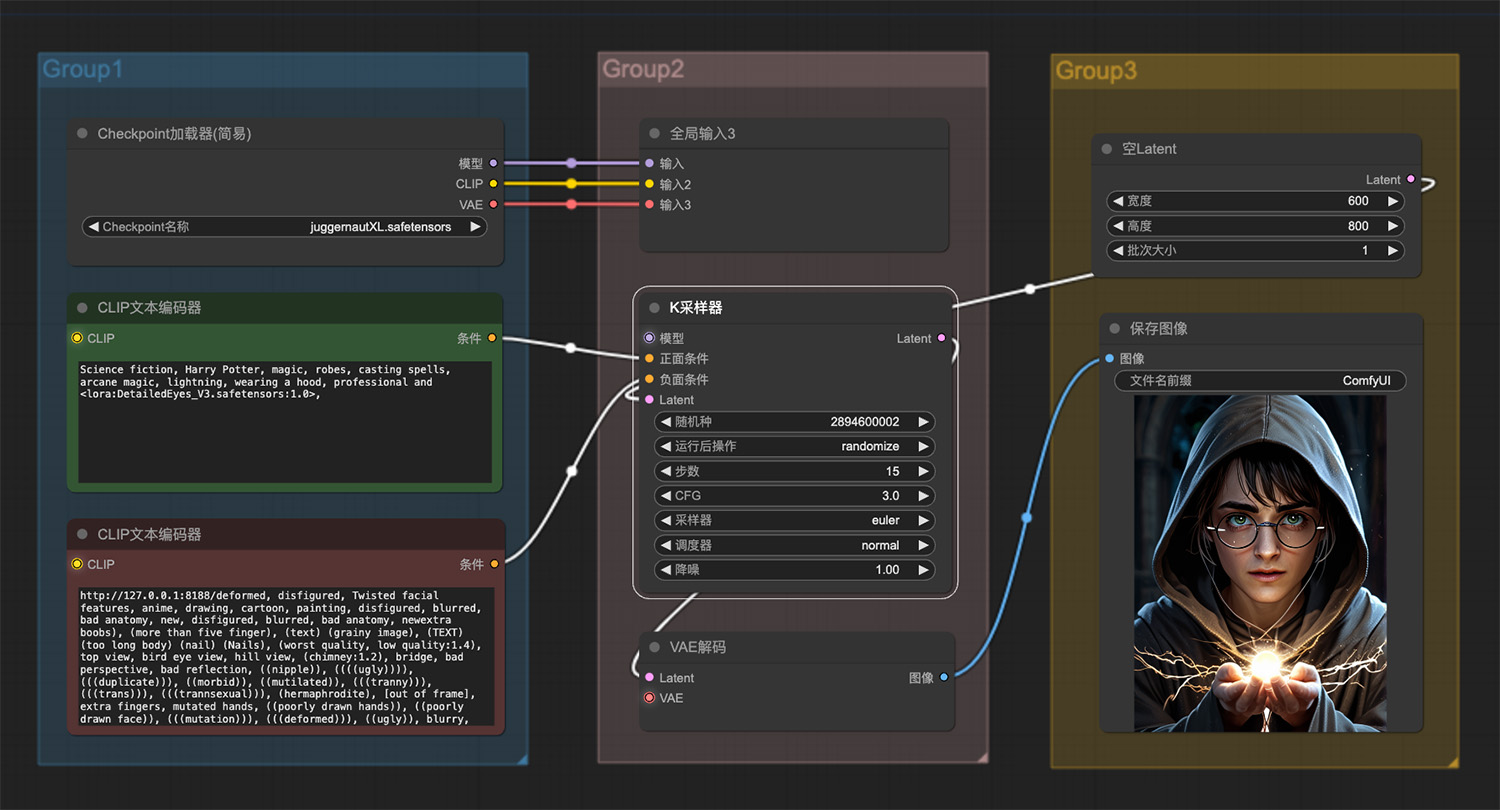 哈利波特魔法ComfyUI工作流
哈利波特魔法ComfyUI工作流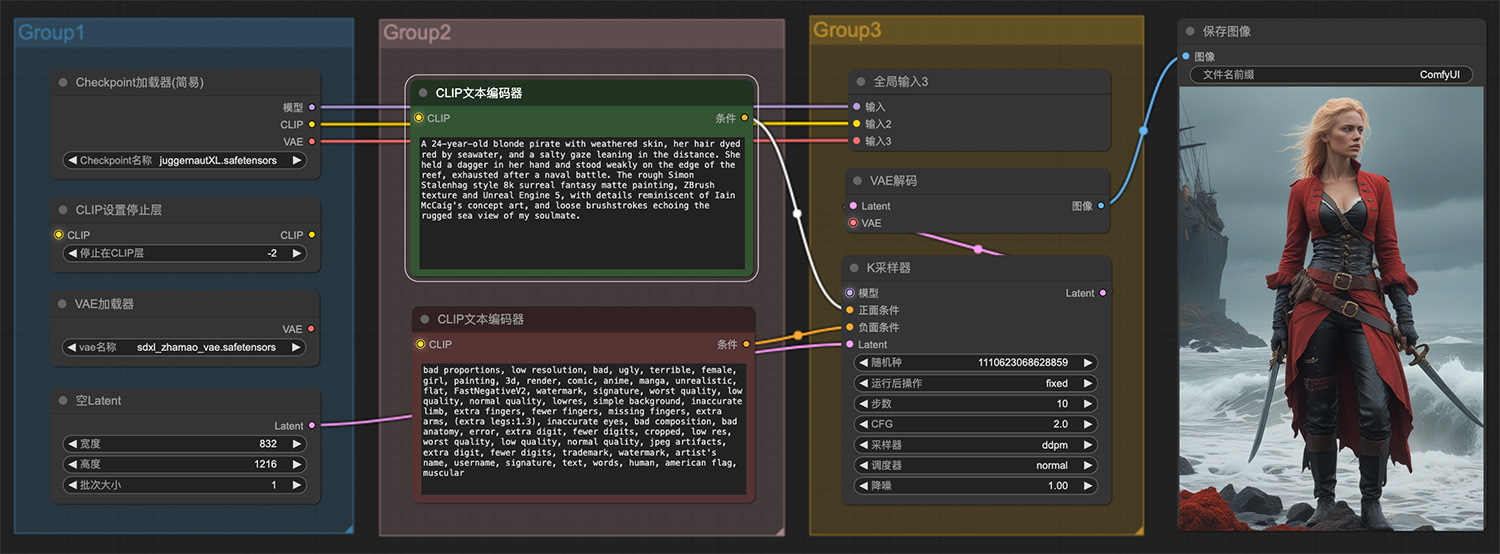 一位24岁的金发女海盗ComfyUI工作流
一位24岁的金发女海盗ComfyUI工作流 一朵在晨光中带着露珠的蓝玫瑰
一朵在晨光中带着露珠的蓝玫瑰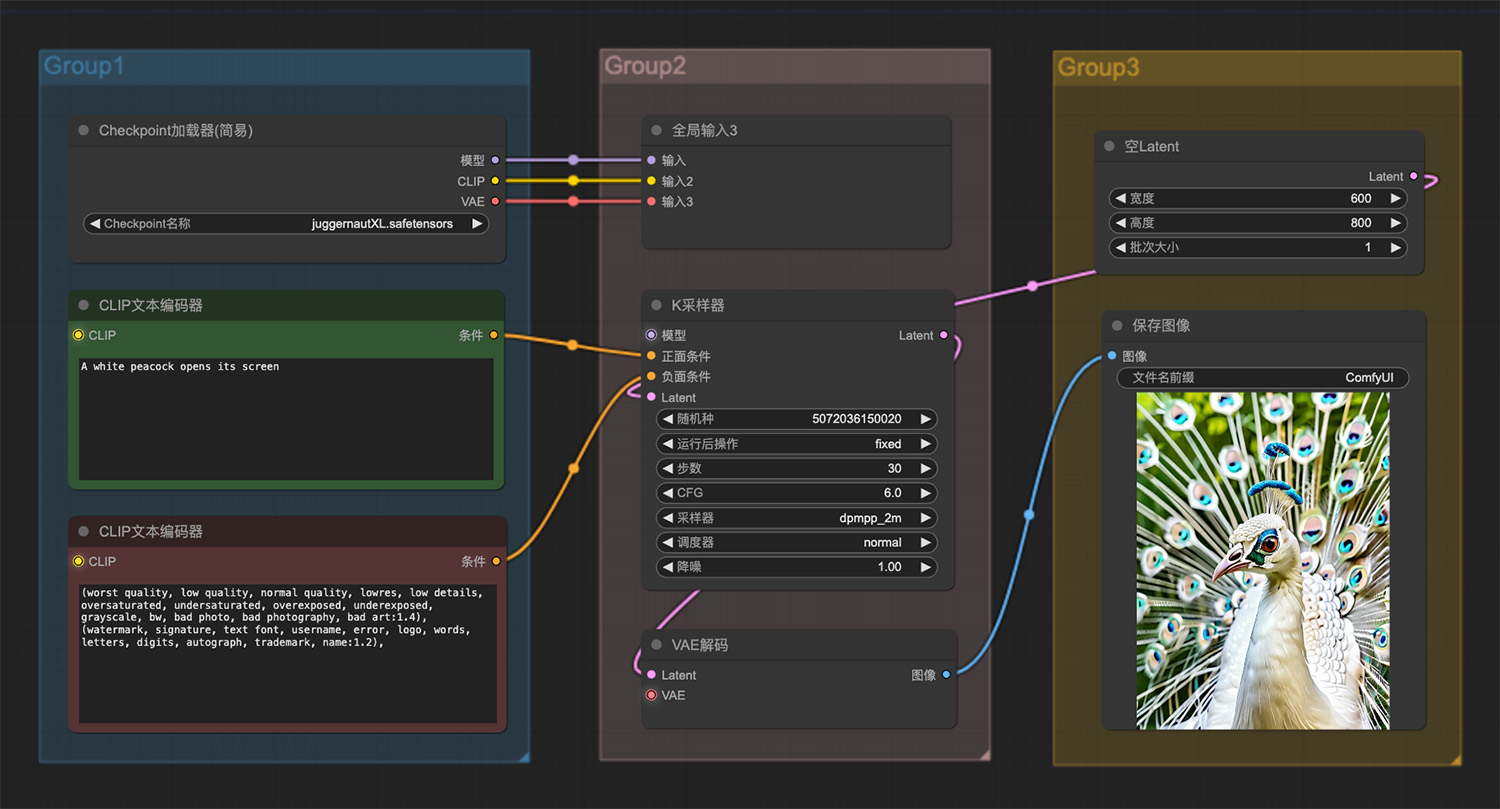 一只开屏的白色孔雀ComfyUI工作流
一只开屏的白色孔雀ComfyUI工作流 即梦AI绘画网页版
即梦AI绘画网页版 woebot
woebot Cradle.bio
Cradle.bio 达医智影
达医智影 Bevel APP最新版
Bevel APP最新版 Lunit
Lunit Profluent.bio
Profluent.bio Antiverse
Antiverse

 豆包在线网页版
豆包在线网页版 GLASS
GLASS 强脑科技
强脑科技